ベイス統計における単回帰分析
頻度主義とベイズ統計のそれぞれの単回帰分析を行いました。
- これからベイス統計をやってみたい人
- StanとRを使って何か始めたい人
向けです。
参考にした本
頻度主義による単回帰分析
頻度主義とは、一般的に単回帰分析と言う場合の手法である。真の母数(パラメータ)があって、それを得られた実データを通して推定するやり方である。真のパラメータを推定する方法として、最尤推定、つまり尤度が最大になるようなパラメータを求める。
ノイズとして正規分布を仮定すると、最尤推定による方法は、最小二乗法と一致する。
> data <- mtcars
> head(data)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1まずは散布図を書く。mpgでdispは負の相関がありそうで、これを単回帰分析してみる。
> library(ggplot2)
> library(GGally)
> ggpairs(data[c(1:3)])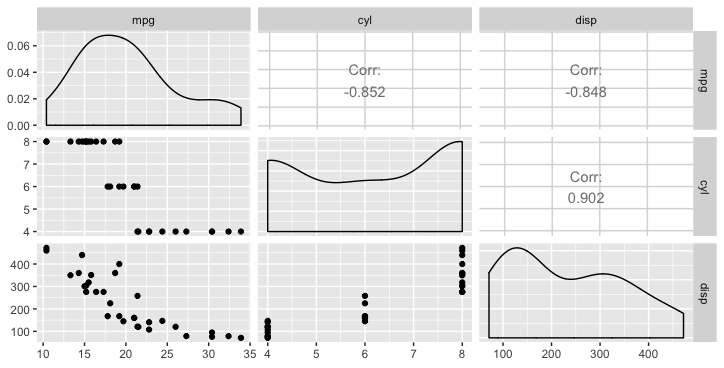
回帰分析はRで非常に簡単に行うことができる。頻度主義による単回帰分析では、切片と傾きが推定するパラメータとなる。下の結果から、mpgに対する傾きは-17.429で、P値も非常に小さい。
> res_lm <- lm(disp ~ mpg, data)
> summary(res_lm)
Call:
lm(formula = disp ~ mpg, data = data)
Residuals:
Min 1Q Median 3Q Max
-103.05 -45.74 -8.17 46.65 153.75
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 580.884 41.740 13.917 1.26e-14 ***
mpg -17.429 1.993 -8.747 9.38e-10 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 66.86 on 30 degrees of freedom
Multiple R-squared: 0.7183, Adjusted R-squared: 0.709
F-statistic: 76.51 on 1 and 30 DF, p-value: 9.38e-10予測区間をプロットすると以下のようになる。下には信頼区間のデータフレームも作成。
> mpg_new <- data.frame(mpg=5:40)
> conf_95 <- predict(res_lm, mpg_new, interval='confidence', level=0.95)
> conf_95 <- data.frame(mpg_new, conf_95)
> pred_95 <- predict(res_lm, mpg_new, interval='prediction', level=0.95)
> pred_95 <- data.frame(mpg_new, pred_95)
> conf_50 <- predict(res_lm, mpg_new, interval='confidence', level=0.50)
> conf_50 <- data.frame(mpg_new, conf_50)
> pred_50 <- predict(res_lm, mpg_new, interval='prediction', level=0.50)
> pred_50 <- data.frame(mpg_new, pred_50)
> p <- ggplot() +
+ geom_ribbon(data=pred_95, aes(x=mpg, ymin=lwr, ymax=upr), alpha=1/6) +
+ geom_ribbon(data=pred_50, aes(x=mpg, ymin=lwr, ymax=upr), alpha=2/6) +
+ geom_point(data=data, aes(x=mpg, y=disp), shape=1, size=5) +
+ geom_line(data=pred_50, aes(x = mpg, y=fit), size=1)
> p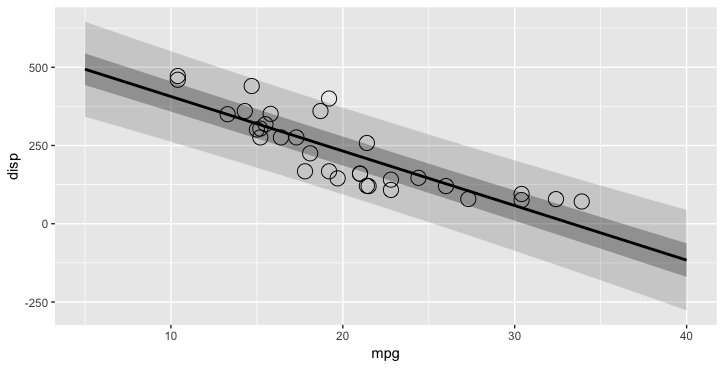
ベイズ統計による単回帰分析
パラメータを確率変数として、その確率分布を求める方法である。データが与えられた時のパラメータ(この場合切片と傾きとノイズ項である正規分布の標準偏差)の確率分布である事後分布を求める。事後分布は尤度関数と事前分布の積に比例する。事後分布を解析的に求める代わりに、尤度関数と事前分布の積からの乱数サンプルを発生させて、事後分布の確率分布を求める。(マルコフ連鎖モンテカルロ法:MCMC)
StanとそのRのパッケージrstanを用いることでこれを簡単に実現できる。
> library(rstan)
> library(ggplot2)
> d <- mtcars
> X_new <- 5:40
> data <- list(N=nrow(d), X=d$mpg, Y=d$disp, N_new=length(X_new), X_new=X_new)ここでdataはデータをrstanモデルに渡すためのインターフェースになる。実データXとYを渡すのはモデル作成に必要なのは当然として、新たなX_newに対する予測値もstan側で計算できる。
次にstanモデルファイルを作成、下記のようになる。事前確率は、無情報事前分布として指定しない。
data {
int N;
real X[N];
real Y[N];
int N_new;
real X_new[N_new];
}
parameters {
real a;
real b;
real<lower=0> sigma;
}
transformed parameters {
real y_base[N];
for (n in 1:N)
y_base[n] = a + b * X[n];
}
model {
for (n in 1:N)
Y[n] ~ normal(a + b*X[n], sigma);
}
generated quantities {
real y_base_new[N_new];
real y_new[N_new];
for (n in 1:N_new) {
y_base_new[n] = a + b * X_new[n];
y_new[n] = normal_rng(y_base_new[n], sigma);
}
}次に実行していく。
> stanmodel <- stan_model(file='model/stan_model_gen.stan')
> fit <- sampling(
+ stanmodel,
+ data=data,
+ init=function() {
+ list(a=runif(1,-10,10), b=runif(1,0,10), sigma=10)
+ },
+ seed=1234,
+ chains = 4, iter = 1000, warmup=200, thin=2
+ )summary(fit)で各パラメータの平均・標準偏差・quantileが確認できる。
> summary(fit)$summary[1:3,]
mean se_mean sd 2.5% 25% 50% 75% 97.5%
a 580.64605 1.64718267 44.941920 489.55591 550.84505 580.97156 608.79399 674.98920
b -17.40673 0.07826705 2.129865 -21.50982 -18.77598 -17.40987 -15.99211 -13.25749
sigma 70.21658 0.28515275 9.796146 54.33300 63.44965 69.08561 75.85365 92.45533
n_eff Rhat
a 744.4229 1.0020684
b 740.5362 1.0011071
sigma 1180.1994 0.9996721傾きbの平均値は-17.40673で、頻度主義における単回帰分析の結果と非常に近いことがわかる。ベイズ的なやり方のメリットとして、傾きbの確率分布(平均と標準偏差など)を把握でき理解しやすい。
ms <- rstan::extract(fit)
data.frame.quantile.mcmc <- function(x, y_mcmc, probs=c(2.5, 25, 50, 75, 97.5)/100) {
qua <- apply(y_mcmc, 2, quantile, probs=probs)
d <- data.frame(X=x, t(qua))
colnames(d) <- c('X', paste0('p', probs*100))
return(d)
}
ggplot.5quantile <- function(data) {
p <- ggplot(data=data)
p <- p + theme_bw(base_size=18)
p <- p + geom_ribbon(aes(x=X, ymin=p2.5, ymax=p97.5), fill='black', alpha=1/6)
p <- p + geom_ribbon(aes(x=X, ymin=p25, ymax=p75), fill='black', alpha=2/6)
p <- p + geom_line(aes(x=X, y=p50), size=1)
return(p)
}
> d_est <- data.frame.quantile.mcmc(x=X_new, y_mcmc=ms$y_new)
> p <- ggplot.5quantile(data=d_est)
> p <- p + geom_point(data=d, aes(x=mpg, y=disp), shape=1, size=5)
> p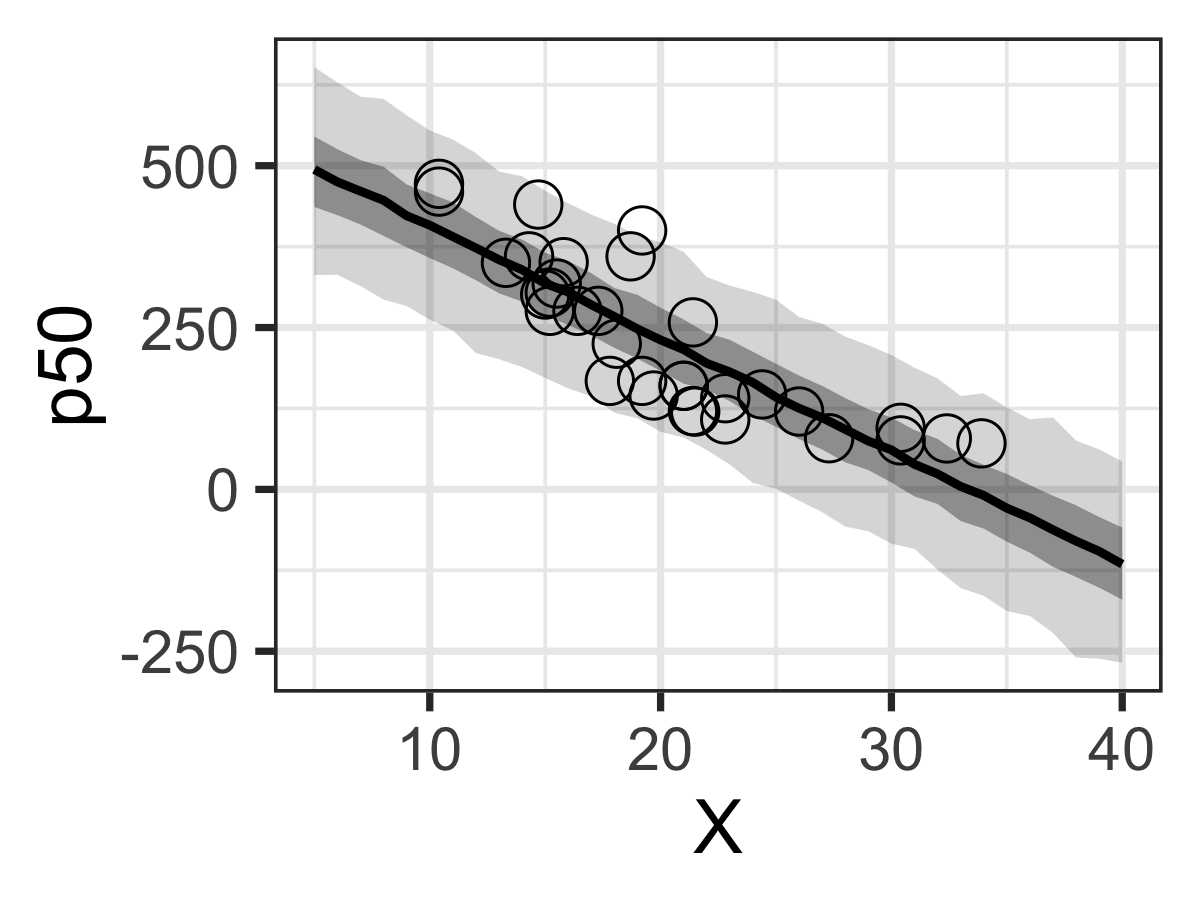
役に立った方はtwitterなどでフォローください。




2019年8月16日
[…] 参考リンク ベイズ統計で単回帰分析 […]